Caracterización molecular de pacientes con cáncer colorrectal
Resumen
Introducción. El cáncer colorrectal tiene una alta incidencia en la población mundial. Diversas vías moleculares están involucradas en su desarrollo, entre ellas, la inestabilidad cromosómica, la inestabilidad microsatelital y la epigenética.
Objetivo. Hacer la caracterización molecular de 44 individuos con cáncer colorrectal esporádico.
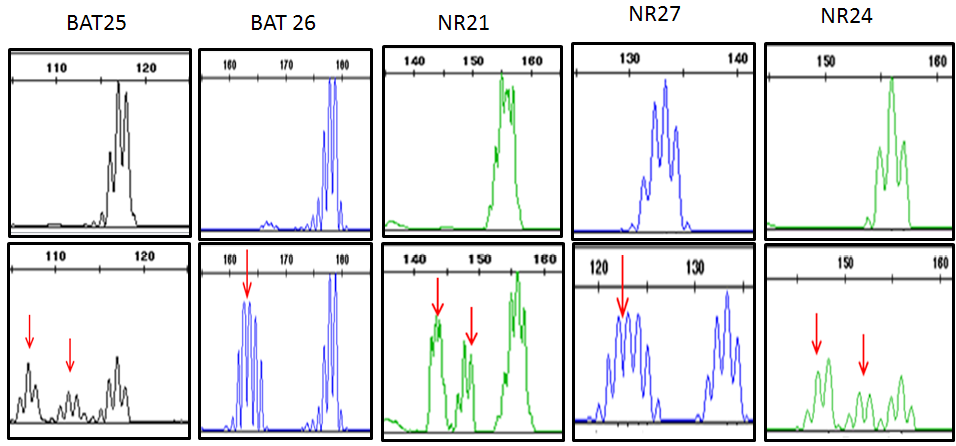
Materiales y métodos. El análisis de mutaciones en los genes APC, KRAS, TP53 y BRAF se hizo mediante secuenciación de Sanger; la inestabilidad microsatelital se determinó mediante electroforesis capilar utilizando cinco marcadores de repetición corta en tándem (Short Tandem Repeat) y el estado de metilación del promotor del gen MLH1 se hizo con la técnica MS-PCR (Methylation-Specific PCR).
Resultados. La frecuencia de mutación de los genes APC, KRAS y TP53 fue del 18,1, 25 y 4,5 %, respectivamente; las mutaciones detectadas se localizaron con mayor frecuencia en el colon derecho. La frecuencia de inestabilidad microsatelital fue del 27,2 % y el 73,1 % en los tumores con metilación en el gen MHL1, y el 91,6 % de los tumores con inestabilidad microsatelital presentaba metilación en el gen MLH1. En el grupo de tumores con estabilidad microsatelital, las mutaciones en los genes APC, KRAS y TP53 fueron más frecuentes que en el grupo de tumores con inestabilidad microsatelital. La metilación del gen MLH1 fue la alteración más predominante.
Conclusiones. En los pacientes con cáncer colorrectal evaluados se demostró la presencia de alteraciones moleculares en las diferentes vías genéticas, las cuales son comunes en su carcinogénesis. Los pacientes presentaron un perfil de mutaciones diferente al de otras poblaciones. Los hallazgos obtenidos en este estudio confirman la heterogeneidad molecular descrita en el desarrollo del cáncer colorrectal.
Descargas
Referencias bibliográficas
Siegel RL, Miller KD, Jemal A. Cancer statistics, 2020. CA Cancer J Clin. 2020;70:7-30. https://doi.org/10.3322/caac.21590
Ferlay J, Colombet M, Soerjomataram I, Mathers C, Parkin DM, Piñeros M, et al. Estimating the global cancer incidence and mortality in 2018: GLOBOCAN sources and methods. Int J Cancer. 2019;15:1941-53. https://doi.org/10.1002/ijc.31937
Siegel RL, Miller KD, Goding Sauer A, Fedewa SA, Butterly LF, Anderson JC, et al. Colorectal cancer statistics, 2020. CA Cancer J Clin. 2020;70:145-64. https://doi.org/10.3322/caac.21601
Dekker E, Tanis PJ, Vleugels JLA, Kasi PM, Wallace MB. Colorectal cancer. Lancet. 2019;394:1467-80. https://doi.org/10.1016/S0140-6736(19)32319-0
Lynch HT, Snyder CL, Shaw TG, Heinen CD, Hitchins MP. Milestones of Lynch syndrome: 1895-2015. Nat Rev Cancer. 2015;15:181-94. https://doi.org/10.1038/nrc3878
Mármol I, Sánchez-de-Diego C, Pradilla-Dieste A, Cerrada E, Rodríguez-Yoldi MJ. Colorectal carcinoma: A general overview and future perspectives in colorectal cancer. Int J Mol Sci. 2017;18:197. https://doi.org/10.3390/ijms18010197
Goel A, Boland CR. Epigenetics of colorectal cancer. Gastroenterology. 2012;143:1442-60. https://doi.org/10.1053/j.gastro.2012.09.032
Fearon ER, Vogelstein B. A genetic model for colorectal tumorigenesis. Cell. 1990;61:759-67. https://doi.org/10.1016/0092-8674(90)90186-i
Worthley DL, Whitehall VL, Spring KJ, Leggett BA. Colorectal carcinogenesis: Road maps to cancer. World J Gastroenterol. 2007;13:3784-91. https://doi.org/10.3748/wjg.v13.i28.3784
Setaffy L, Langner C. Microsatellite instability in colorectal cancer: Clinicopathological significance. Pol J Pathol. 2015;66:203-18. https://doi.org/10.5114/pjp.2015.54953
Suraweera N, Duval A, Reperant M, Vaury C, Furlan D, Leroy K, et al. Evaluation of tumor microsatellite instability using five quasimonomorphic mononucleotide repeats and pentaplex PCR. Gastroenterology. 2002;123:1804-11. https://doi.org/10.1053/gast.2002.37070
Jia Y, Guo M. Epigenetic changes in colorectal cancer. Chin J Cancer. 2013;32:21-30. https://doi.org/10.5732/cjc.011.10245
Li X, Yao X, Wang Y, Hu F, Wang F, Jiang L, et al. MLH1 promoter methylation frequency in colorectal cancer patients and related clinicopathological and molecular features. PLoS ONE. 2013;8:e59064. https://doi.org/10.1371/journal.pone.0059064
Cancer Genome Atlas Network. Comprehensive molecular characterization of human colon and rectal cancer. Nature. 2012;487:330-7. https://doi.org/10.1038/nature11252
Lee MS, Menter DG, Kopetz S. Right versus left colon cancer biology: Integrating the consensus molecular subtypes. J Natl Compr Canc Netw. 2017;15:411-9. https://doi.org/10.6004/jnccn.2017.0038
Guinney J, Dienstmann R, Wang X, de Reyniès A, Schlicker A, Soneson C, et al. The consensus molecular subtypes of colorectal cancer. Nat Med. 2015;21:1350-6. https://doi.org/10.1038/nm.3967
Peeters M, Douillard JY, van Cutsem E, Siena S, Zhang K, Williams R, et al. Mutant KRAS codon 12 and 13 alleles in patients with metastatic colorectal cancer: Assessment as prognostic and predictive biomarkers of response to panitumumab. J Clin Oncol. 2013;20;31:759-65. https://doi.org/10.1200/JCO.2012.45.1492
Nguyen HT, Duong HQ. The molecular characteristics of colorectal cancer: Implications for diagnosis and therapy. Oncol Lett. 2018;16:9-18. https://doi.org/10.3892/ol.2018.8679
Murcia O, Juárez M, Rodríguez-Soler M, Hernández-Illán E, Giner-Calabuig M, Alustiza M, et al. Colorectal cancer molecular classification using BRAF, KRAS, microsatellite instability and CIMP status: Prognostic implications and response to chemotherapy. PLoS ONE. 2018;6:13:e0203051. https://doi.org/10.1371/journal.pone.0203051
Kawakami H, Zaanan A, Sinicrope FA. Microsatellite instability testing and its role in the management of colorectal cancer. Curr Treat Options Oncol. 2015;16:30-44. https://doi.org/10.1007/s11864-015-0348-2
Smith G, Carey FA, Beattie J, Wilkie MJ, Lightfoot TJ, Coxhead J, et al. Mutations in APC, Kirsten-ras, and p53--alternative genetic pathways to colorectal cancer. Proc Natl Acad Sci USA. 2002;99:9433-8. https://doi.org/10.1073/pnas.122612899
Vasovcak P, Pavlikova K, Sedlacek Z, Skapa P, Kouda M, Hoch J, et al. Molecular genetic analysis of 103 sporadic colorectal tumours in Czech patients. PLoS ONE. 2011;6:e24114. https://doi.org/10.1371/journal.pone.0024114
Palacio-Rúa KA, Isaza-Jiménez LF, Ahumada-Rodríguez E, Muñetón-Peña CM. Genetic analysis in APC, KRAS, and TP53 in patients with stomach and colon cancer. Rev Gastroenterol Mex. 2014;79-89. https://doi.org/10.1016/j.rgmx.2014.05.001
Al-Shamsi HO, Jones J, Fahmawi Y, Dahbour I, Tabash A, Abdel-Wahab R, et al. Molecular spectrum of KRAS, NRAS, BRAF, PIK3CA, TP53, and APC somatic gene mutations in Arab patients with colorectal cancer: Determination of frequency and distribution pattern. J Gastrointest Oncol. 2016;7:882-902. https://doi.org/10.21037/jgo.2016.11.02
Syed Sameer A, Shah ZA, Abdullah S, Chowdri NA, Siddiqi MA. Analysis of molecular aberrations of Wnt pathway gladiators in colorectal cancer in the Kashmiri population. Hum Genomics. 2011;5:441-52. https://doi.org/10.1186/1479-7364-5-5-441
Kapitanović S, Cacev T, Radosević S, Spaventi S, Spaventi R, Pavelić K. APC gene loss of heterozygosity, mutations, E1317Q, and I1307K germ-line variants in sporadic colon cancer in Croatia. Exp Mol Pathol. 2004;77:193-200. https://doi.org/10.1016/j.yexmp.2004.06.001
Michailidi C, Theocharis S, Tsourouflis G, Pletsa V, Kouraklis G, Patsouris E, et al. Expression and promoter methylation status of hMLH1, MGMT, APC, and CDH1 genes in patients with colon adenocarcinoma. Exp Biol Med (Maywood). 2015;240:1599-605. https://doi.org/10.1177/1535370215583800
Zhang J, Zheng J, Yang Y, Lu J, Gao J, Lu T, et al. Molecular spectrum of KRAS, NRAS, BRAF and PIK3CA mutations in Chinese colorectal cancer patients: Analysis of 1,110 cases. Sci Rep. 2015;5:18678. https://doi.org/10.1038/srep18678
Guo F, Gong H, Zhao H, Chen J, Zhang Y, Zhang L, et al. Mutation status and prognostic values of KRAS, NRAS, BRAF and PIK3CA in 353 Chinese colorectal cancer patients. Sci Rep. 2018;8:6076. https://doi.org/10.1038/s41598-018-24306-1
Zlobec I, Kovac M, Erzberger P, Molinari F, Bihl MP, Rufle A, et al. Combined analysis of specific KRAS mutation, BRAF and microsatellite instability identifies prognostic subgroups of sporadic and hereditary colorectal cancer. Int J Cancer. 2010;127:2569-75. https://doi.org/10.1002/ijc.25265
Conlin A, Smith G, Carey FA, Wolf CR, Steele RJ. The prognostic significance of K-ras, p53, and APC mutations in colorectal carcinoma. Gut. 2005;54:1283-6. https://doi.org/10.1136/gut.2005.066514
Cai ZX, Tang XD, Gao HL, Tang C, Nandakumar V, Jones L, et al. APC, FBXW7, KRAS, PIK3CA, and TP53 gene mutations in human colorectal cancer tumors frequently detected by next-generation DNA sequencing. J Mol Genet Med. 2014;8:145-56. https://doi.org/10.4172/1747-0862.1000145
Hainaut P, Pfeifer GP. Somatic TP53 mutations in the era of genome sequencing. Cold Spring Harb Perspect Med. 2016;6:a026179. https://doi.org/10.1101/cshperspect.a026179
Naccarati A, Polakova V, Pardini B, Vodickova L, Hemminki K, Kumar R, et al. Mutations and polymorphisms in TP53 gene--an overview on the role in colorectal cancer. Mutagenesis. 2012;27:211-8. https://doi.org/10.1093/mutage/ger067
Herrera JC, Isaza LF, Ramírez JL, Vásquez G, Muñetón CM. Detección de aneuploidías del cromosoma 17 y deleción del gen TP53 en una amplia variedad de tumores sólidos mediante hibridación in situ fluorescente bicolor. Biomédica. 2010;30:390-0. https://doi.org/10.7705/biomedica.v30i3.273
Ramírez G, Herrera J, Muñetón CM, Márquez JR, Isaza LF. Análisis de las aneuploidías del cromosoma 17 y deleción del gen TP53 en tumores gastrointestinales por FISH-bicolor. Rev Col Gastroenterol. 2008;23:333-42. https://doi.org/10.22516/issn.2500-7440
Scott RJ, Crooks R, Rose L, Attia J, Thakkinstian A, Thomas L, et al. Germline missense changes in the APC gene and their relationship to disease. Hered Cancer Clin Pract. 2004;2:81-91. https://doi.org/10.1186/1897-4287-2-2-81
Fernández-Rozadilla C, de Castro L, Clofent J, Brea-Fernández A, Bessa X, Abulí A, et al. Gastrointestinal Oncology Group of the Spanish Gastroenterological Association. Single nucleotide polymorphisms in the Wnt and BMP pathways and colorectal cancer risk in a Spanish cohort. PLoS ONE. 2010;5:e12673. https://doi.org/10.1371/journal.pone.0012673
Wachsmannova-Matelova L, Stevurkova V, Adamcikova Z, Holec V, Zajac V. Polymorphisms in the adenomatous polyposis coli gene in Slovak families suspected of FAP. Neuro Endocrinol Lett. 2009;30:25-8.
Bae JM, Kim JH, Kang GH. Molecular subtypes of colorectal cancer and their clinicopathologic features, with an emphasis on the serrated neoplasia pathway. Arch Pathol Lab Med. 2016;140:406-12. https://doi.org/10.5858/arpa.2015-0310-RA
Montenegro Y, Ramírez-Castro JL, Isaza LF, Bedoya G, Muñetón-Peña CM. Microsatellite instability among patients with colorectal cancer. Rev Med Chil. 2006;134:1221-9. https://doi.org/ 10.4067/s0034-98872006001000002
Phipps AI, Lindor NM, Jenkins MA, Baron JA, Win AK, Gallinger S, et al. Colon and rectal cancer survival by tumor location and microsatellite instability: The Colon Cancer Family Registry. Dis Colon Rectum. 2013;56:937-44. https://doi.org/10.1097/DCR.0b013e31828f9a57
Wielandt AM, Hurtado C, Moreno C M, Villarroel C, Castro M, Estay M, et al. Characterization of Chilean patients with sporadic colorectal cancer according to the three main carcinogenic pathways: Microsatellite instability, CpG island methylator phenotype and chromosomal instability. Tumour Biol. 2020;42:1010428320938492.
https://doi.org/10.1177/1010428320938492
Clarke CN, Kopetz ES. BRAF mutant colorectal cancer as a distinct subset of colorectal cancer: Clinical characteristics, clinical behavior, and response to targeted therapies. J Gastrointest Oncol. 2015;6:660-7. https://doi.org/10.3978/j.issn.2078-6891.2015.077
Siddique S, Tariq K, Rafiq S, Raheem A, Ahmed R, Shabbir-Moosajee M, et al. Sporadic early onset colorectal cancer in Pakistan: A case- control analysis of microsatellite instability. Asian Pac J Cancer Prev. 2016;17:2587-92. https://doi.org/10.7314/APJCP.2016.17.5.2587
Agostini M, Enzo MV, Morandi L, Bedin C, Pizzini S, Mason S, et al. A ten markers panel provides a more accurate and complete microsatellite instability analysis in mismatch repairdeficient colorectal tumors. Cancer Biomark. 2010;6:49-61. https://doi.org/10.3233/CBM-2009-0118
Hu J, Yan WY, Xie L, Cheng L, Yang M, Li L, et al. Coexistence of MSI with KRAS mutation is associated with worse prognosis in colorectal cancer. Medicine. 2016;95:e5649. https://doi.org/10.1097/MD.0000000000005649
Li X, Yao X, Wang Y, Hu F, Wang F, Jiang L, et al. MLH1 promoter methylation frequency in colorectal cancer patients and related clinicopathological and molecular features. PLoS ONE. 2013;8:e59064. https://doi.org/ 10.1371/journal.pone.0059064
de Vogel S, Weijenberg MP, Herman JG, Wouters KA, de Goeij AF, van den Brandt PA, et al. MGMT and MLH1 promoter methylation versus APC, KRAS and BRAF gene mutations in colorectal cancer: Indications for distinct pathways and sequence of events. Ann Oncol. 2009;20:1216-22. https://doi.org/ 10.1093/annonc/mdn782
Miladi-Abdennadher I, Abdelmaksoud-Damak R, Ayadi L, Khabir A, Frikha F, Kallel L, et al. Aberrant methylation of hMLH1 and p16INK4a in Tunisian patients with sporadic colorectal adenocarcinoma. Biosci Rep. 2011;31:257-64. https://doi.org/10.1042/BSR20100023
Poynter JN, Siegmund KD, Weisenberger DJ, Long TI, Thibodeau SN, Lindor N, et al. Colon Cancer Family Registry Investigators. Molecular characterization of MSI-H colorectal cancer by MLHI promoter methylation, immunohistochemistry, and mismatch repair germline mutation screening. Cancer Epidemiol Biomarkers Prev. 2008;17:3208-15. https://doi.org/10.1158/1055-9965.EPI-08-0512
Algunos artículos similares:
- Oscar F. Herrán, María F. Ardila, Martha P. Rojas, Gustavo A. Hernández, Diseño de cuestionarios de frecuencia de consumo para estudiar la relación dieta-cáncer en Colombia , Biomédica: Vol. 30 Núm. 1 (2010)
- Brian Alejandro Suárez, Claudia Liliana Cuervo, Concepción Judith Puerta, La región intergénica del gen H2A apoya las subpoblaciones KP1(-) y KP1(+) de Trypanosoma rangeli , Biomédica: Vol. 27 Núm. 3 (2007)
- Patricia Escandón, Popchai Ngamskulrungroj, Wieland Meyer, Elizabeth Castañeda, Determinación in vitro de la pareja sexual en aislamientos del complejo Cryptococcus neoformans , Biomédica: Vol. 27 Núm. 2 (2007)
- Juan Carlos Cataño, Síndrome miasténico de Eaton-Lambert , Biomédica: Vol. 30 Núm. 3 (2010)
- Juan Carlos Herrera, Luis Fernando Isaza, José Luis Ramírez, Gonzalo Vásquez, Carlos Mario Muñetón, Detección de aneuploidías del cromosoma 17 y deleción del gen TP53 en una amplia variedad de tumores sólidos mediante hibridación in situ fluorescente bicolor , Biomédica: Vol. 30 Núm. 3 (2010)
- Gabriel Bedoya, Jenny García, Patricia Montoya, Winston Rojas, Maria Eugenia Amézquita, Iván Soto, Maria Cecilia López, Jorge Ospina-Duque, Andrés Ruiz-Linares, Análisis de isonimia entre poblaciones del noroeste de Colombia , Biomédica: Vol. 26 Núm. 4 (2006)
- Yaliana Tafurt-Cardona, Leidy D. Jaramillo-Ruiz, Wilson Muñoz-Ordóñez, Sulma L. Muñoz-Benítez, Carlos H. Sierra-Torres, Mayor frecuencia de aberraciones cromosómicas en linfocitos expuestos o no a mitomicina C, de mujeres posmenopáusicas obesas en comparación con mujeres no obesas del departamento del Cauca, Colombia , Biomédica: Vol. 32 Núm. 3 (2012)
- Luis Caraballo, Josefina Zakzuk, Consideraciones sobre la evolución de la respuesta inmunitaria Th2 y sus posibles relaciones con parasitosis y alergia , Biomédica: Vol. 32 Núm. 1 (2012)
- Ricardo Cendales, Constanza Pardo, Claudia Uribe, Guillermo López, María Clara Yépez, Luis Eduardo Bravo, Calidad de los datos en los registros de cáncer de base de población en Colombia , Biomédica: Vol. 32 Núm. 4 (2012)
- Lisseth Cabarcas, Eugenia Espinosa, Harvy Velasco, Etiología del retardo mental en la infancia: experiencia en dos centros de tercer nivel , Biomédica: Vol. 33 Núm. 3 (2013)
| Estadísticas de artículo | |
|---|---|
| Vistas de resúmenes | |
| Vistas de PDF | |
| Descargas de PDF | |
| Vistas de HTML | |
| Otras vistas | |
Datos de los fondos
-
Universidad de Antioquia
Números de la subvención CODI-CPT- 1231.

























